ساختارهای dna
پاسخ : ساختارهای dna
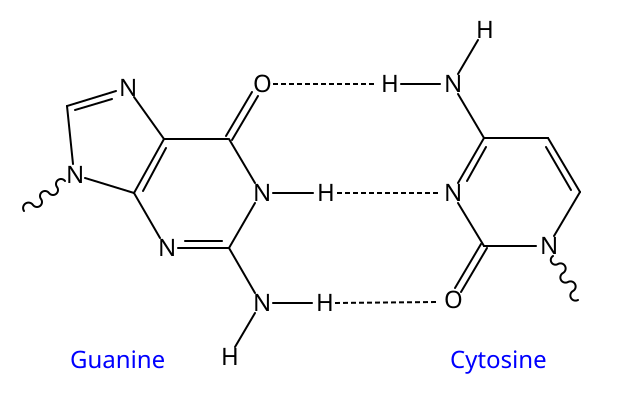
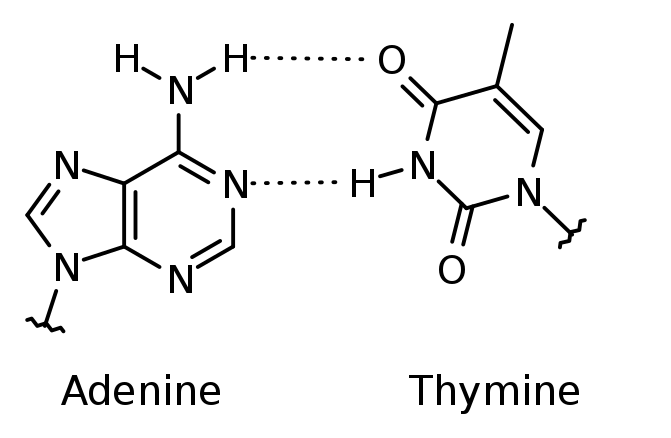
در سال ۱۹۴۸ لینوس پاولینگ کشف کرد که بسیاری از مولکولهای پروتئینی به شکل یک مارپیچ هستند، و کم و بیش شکلی همانند فنر دارند. در سال ۱۹۵۰ نیز اروین شارگاف نشان داد که اگرچه آرایش بازهای موجود در ساختار DNA بسیار گوناگون است، ولی همواره نسبت باز آدنین و باز تیمین موجود در آن با هم برابر است و همین طور نسبت باز سیتوزین با باز گوانین. این دو یافته نقش مهمی را در آشکار شدن ساختار مولکول DNA داشتند. در دهه ۱۹۵۰ همچنان رقابت برای یافتن ساختار DNA ادامه داشت. در دانشگاه کمبریج فرانسیس کریک و جیمز واتسون برپایه کارهای پاولینگ کوشش داشتند تا با ارایه مدلهای فیزیکی ساختارهای احتمالی ممکن برای DNA را محدود کنند تا سرانجام به ساختار درست دست یابند. گروه دیگری در برگیرنده موریس ویلکینز و رزالین فرانکلین نیز در کالج کینگ لندن همزمان سرگرم مطالعه DNA بود. روش کار این گروه با گروه پیشین متفاوت بود. آنها کوشش داشتند تا با روش آزمایشگاهی به ویژه با بکارگیری تصاویر پراش اشعه X از مولکول DNA، ساختار آن را معین کنند.
در سال ۱۹۵۱، فرانکلین دریافت که DNA با نگرش به میزان نم هوای پیرامون، میتواند دو شکل متفاوت داشته باشد و بنابراین نتیجه گیری کرد که بخش فسفات مولکول در سمت بیرونی آن قرار دارد. اندکی بعد او با بکارگیری تصاویر اشعه X فهمید که DNA در حالت «نمناک» از همگی ویژگیهای یک مارپیچ برخوردار است؛ این احتمال که حالت دیگر مولکول DNA نیز به شکل مارپیچی باشد به ذهن او خطور کرد، ولی نمیخواست تا زمانی که شواهد پایانی برای این حدس پیدا کند آن را اعلام نماید. در ژانویه ۱۹۵۳ ویلکینز که از به نتیجه رسیدن تحقیقات ناامید شده بود، نتایج تحقیقات فرانکلین را بدون اطلاع و خوشنودی او، با واتسون در میان گذاشت. واتسون و کریک با بکارگیری این نتایج مدلی بسیار شگفت انگیز را برای ساختار DNA پیشنهاد نمودند. آنها مولکول را به گونه دو زنجیر مارپیچی در برگیرنده نوکلئوتیدها تصور کردند که یکی از آنها بالا میرفت و دیگری پایین میآمد. کریک که به تازگی یافتههای شارگاف را هم مطالعه کرده بود کوشش کرد با بکارگیری آنها روش قرار گرفتن بازها را در مولکول DNA مشخص کند. او اظهار کرد که بازها در میانه این مارپیچ دوتایی دو به دو به هم متصل میشوند تا فاصله میان دو مارپیچ ثابت بماند. آنها ادعا کردند که هر یک از این دو مارپیچ مولکول DNA میتواند به نام قالبی برای ایجاد دیگری بهره گیری شود. در تقسیم سلولی این دو رشته از هم جدا میشوند و بر روی هر یک از آنها یک نمونه جدید همانند رشته مقابل پیشین ساخته میشود. با این روش بدون اینکه ساختار DNA عوض شود، یک DNA همانند آن فرآوری میشود. در اندک مواردی که در این روند خطایی پیش بیاید، گواه «جهش» خواهیم بود. مدل آنها چنان با اطلاعات برآمده از آزمایشها مطابقت داشت که بی درنگ مورد قبول همه واقع شد. کشف ساختار DNA را میتوان مهمترین یافته زیستی در صد سال گذشته دانست. در سال ۱۹۶۲ واتسون، کریک و ویلکینز موفق به دریافت پاداش نوبل شدند، ولی فرانکلین در گذشته بود.
در سال ۱۹۴۸ لینوس پاولینگ کشف کرد که بسیاری از مولکولهای پروتئینی به شکل یک مارپیچ هستند، و کم و بیش شکلی همانند فنر دارند. در سال ۱۹۵۰ نیز اروین شارگاف نشان داد که اگرچه آرایش بازهای موجود در ساختار DNA بسیار گوناگون است، ولی همواره نسبت باز آدنین و باز تیمین موجود در آن با هم برابر است و همین طور نسبت باز سیتوزین با باز گوانین. این دو یافته نقش مهمی را در آشکار شدن ساختار مولکول DNA داشتند. در دهه ۱۹۵۰ همچنان رقابت برای یافتن ساختار DNA ادامه داشت. در دانشگاه کمبریج فرانسیس کریک و جیمز واتسون برپایه کارهای پاولینگ کوشش داشتند تا با ارایه مدلهای فیزیکی ساختارهای احتمالی ممکن برای DNA را محدود کنند تا سرانجام به ساختار درست دست یابند. گروه دیگری در برگیرنده موریس ویلکینز و رزالین فرانکلین نیز در کالج کینگ لندن همزمان سرگرم مطالعه DNA بود. روش کار این گروه با گروه پیشین متفاوت بود. آنها کوشش داشتند تا با روش آزمایشگاهی به ویژه با بکارگیری تصاویر پراش اشعه X از مولکول DNA، ساختار آن را معین کنند.
در سال ۱۹۵۱، فرانکلین دریافت که DNA با نگرش به میزان نم هوای پیرامون، میتواند دو شکل متفاوت داشته باشد و بنابراین نتیجه گیری کرد که بخش فسفات مولکول در سمت بیرونی آن قرار دارد. اندکی بعد او با بکارگیری تصاویر اشعه X فهمید که DNA در حالت «نمناک» از همگی ویژگیهای یک مارپیچ برخوردار است؛ این احتمال که حالت دیگر مولکول DNA نیز به شکل مارپیچی باشد به ذهن او خطور کرد، ولی نمیخواست تا زمانی که شواهد پایانی برای این حدس پیدا کند آن را اعلام نماید. در ژانویه ۱۹۵۳ ویلکینز که از به نتیجه رسیدن تحقیقات ناامید شده بود، نتایج تحقیقات فرانکلین را بدون اطلاع و خوشنودی او، با واتسون در میان گذاشت. واتسون و کریک با بکارگیری این نتایج مدلی بسیار شگفت انگیز را برای ساختار DNA پیشنهاد نمودند. آنها مولکول را به گونه دو زنجیر مارپیچی در برگیرنده نوکلئوتیدها تصور کردند که یکی از آنها بالا میرفت و دیگری پایین میآمد. کریک که به تازگی یافتههای شارگاف را هم مطالعه کرده بود کوشش کرد با بکارگیری آنها روش قرار گرفتن بازها را در مولکول DNA مشخص کند. او اظهار کرد که بازها در میانه این مارپیچ دوتایی دو به دو به هم متصل میشوند تا فاصله میان دو مارپیچ ثابت بماند. آنها ادعا کردند که هر یک از این دو مارپیچ مولکول DNA میتواند به نام قالبی برای ایجاد دیگری بهره گیری شود. در تقسیم سلولی این دو رشته از هم جدا میشوند و بر روی هر یک از آنها یک نمونه جدید همانند رشته مقابل پیشین ساخته میشود. با این روش بدون اینکه ساختار DNA عوض شود، یک DNA همانند آن فرآوری میشود. در اندک مواردی که در این روند خطایی پیش بیاید، گواه «جهش» خواهیم بود. مدل آنها چنان با اطلاعات برآمده از آزمایشها مطابقت داشت که بی درنگ مورد قبول همه واقع شد. کشف ساختار DNA را میتوان مهمترین یافته زیستی در صد سال گذشته دانست. در سال ۱۹۶۲ واتسون، کریک و ویلکینز موفق به دریافت پاداش نوبل شدند، ولی فرانکلین در گذشته بود.
پاسخ : ساختارهای dna
A. DNA شامل زنجیره های پلیمری بلند است .B. DNA حاوی دیوکسی ریبونوکلوزیدهایی در3-5 در پیوند با فسفو دیستر می باشد .C. پیشنهاد فرستادن انکسار اشعه X به ساختار مارپیچ - الگو متقاتع پیشنهاد یک گام مارپیچ با زاویه 45 را داد .- بازتابهای محوری واحد خای تکراری از4/3 تا 34 A را داد .- بازتابهای شعایی یک فیبر نوری با عرض 20A را داد .D. کارشارکاف نشان داد که اساس ترکیب بندی DNA از موجود زنده ای به موجود زنده ای دیگر متفاوت می باشد .اما روابط خاصی بین مقادیر از پایه های مختلف همیشه برگذار می شود . این روابط قوانین شارگاف نام گرفت : - مقدار آدنین برابر با مقدار تیمین است .- مقدار گوانین برابر مقدار سیتین است .- مقدار آدنین به علاوه گوانین معادل 50% از کل می باشد که نشان دهنده این است که 50% از پایه های اصلی DNA پورتین هستند .- مقداری از تیمین به علاوه سیتوزین معادل 50% از کل می باشد که نشان دهنده این است که 50% پایه های DNA (pyrimidines) هستند .E.پیشنهاد واتسون وکریک جفت شدن پایه بود که توضیح دهنده تساوی شارگاف می باشد .- هدف از انتخاب جفت باز اتصال به وسیله پیوند هیدروژنی بود . پایه ها درحالت طبیعی به شکل tautomeric (خالی ) درPH 7 می باشند .- انتخاب یک جفت AU ویک جفت GC یک superimposable در محل ارتبط با glycosylic داد.- یک نتیجه بدست آمده این است که ساختار DNA از یک توالی مستقل تشکیل شده است .- اولین جفت باز مشاهده شده در آزمایش کریتوگرافی اشعه X جفت باز hoogstein بود نه جفت باز واتسون وکریک . F.دی اکسی ریبوز ها در هر جفت از پایه ها در جهت مخالف هم وصل شده اند.G.هر پایه مخالف وبرابر با پایه دیگر می باشد.H.این شکل ترکیب از نوکلئواسیدهادر قطب مخالف DNA زنجیره ای مارپیچ را حاصل می کند.I.این ترکیب ودر نتیجه زنجیره antiparallelتولید یک محور جفت ومتقارن در هریک از جفت ها می نماید (محور تقارن دورانی دو برابر) محور جفت برای پروتئین های متصل به DNA بسیار مهم می باشد. اکثر پروتئینهای متصل شونده به DNA دارای محور تقارن و متصل به DNA وبه صورت متقارن ومتوالی می باشند.J.این ساختار دارای یک جفت پایه به عنوان واحد اولیه تکرار می باشد.ونتایجبه صورت یک تقارن بین هر جفت پایه حول محورهای اضافی می باشد.K.سپس واتسون وکریک جفت بازهایی را با باندهای phosphodiester که فاصله پایه ها از هم A4/3 واز هم جدا بودند را به هم متصل کردند وهر جفت پایه بعدی واز هم جدا بودند را به هم متصل کردند وهر جفت پایه بعدی 36 درجه چرخش داشت.این چرخش تولید یک مارپیچ راست گردان دوبله می نماید که با 10 پایه در هر نوبت ایجاد تکرار عناصر در هر 4/3 وA34 می نماید.L .زاویه منفرجه ایجاد شده از ارتباط گلیکوزیدی منجر به پیدایش شیارهای عمده و جزئی در مارپیچ با گروهای خاصی در هر گروه می گردد.M.پایه ها بر حول محور مارپیچ عمود می باشند.
پاسخ : ساختارهای dna
شکل های قبلی با توضیح
 [TABLE="class: wikitable"]
[TABLE="class: wikitable"]
[TR]
[TH]شکل های مارپیچ DNA[/TH]
[TH]حالت A[/TH]
[TH]حالتB[/TH]
[TH]حالت Z[/TH]
[/TR]
[TR]
[TD]نسبت های کلی[/TD]
[TD]کوتاه و پهن[/TD]
[TD]بزرگتر و باریکتر[/TD]
[TD]طویل و باریک[/TD]
[/TR]
[TR]
[TD]ارتفاع به ازای هرجفت باز[/TD]
[TD]3/2آنگستروم[/TD]
[TD]23/3آنگستروم[/TD]
[TD]8/3آنگستروم[/TD]
[/TR]
[TR]
[TD]قطر مارپیچ[/TD]
[TD]5/25آنگستروم[/TD]
[TD]7/23آنگستروم[/TD]
[TD]4/18آنگستروم[/TD]
[/TR]
[TR]
[TD]جهت چرخش مارپیچ[/TD]
[TD]راست گرد[/TD]
[TD]راست گرد[/TD]
[TD]چپ گرد[/TD]
[/TR]
[TR]
[TD]خمیدگی باز نسبت به محور مارپیچ[/TD]
[TD]19+[/TD]
[TD]2/1-[/TD]
[TD]9-[/TD]
[/TR]
[TR]
[TD]متوسط چرخش پروانه ای جفت باز[/TD]
[TD]18+[/TD]
[TD]16+[/TD]
[TD]حدود 0[/TD]
[/TR]
[TR]
[TD]موقعیت محور مارپیچ[/TD]
[TD]شیار بزرگ[/TD]
[TD]از میان جفت باز ها[/TD]
[TD]شیار کوچک[/TD]
[/TR]
[TR]
[TD]اندازه ی شیار بزرگ[/TD]
[TD]بسیار باریک با عمق زیاد[/TD]
[TD]پهن و عمق متوسط[/TD]
[TD]پهن شده به روی سطح مارپیچ[/TD]
[/TR]
[TR]
[TD]اندازه ی شیار کوچک[/TD]
[TD]بسیار پهن ولی کم عمق[/TD]
[TD]باریک و عمق متوسط[/TD]
[TD]بسیار باریک ولی خیلی عمیق[/TD]
[/TR]
[TR]
[TD]صورت بندی پیوند گلیکوزیدی[/TD]
[TD]آنتی[/TD]
[TD]آنتی[/TD]
[TD]آنتی در Cو سین در G[/TD]
[/TR]
[TR]
[TD]جفت باز در هر دور مارپیچ[/TD]
[TD]11[/TD]
[TD]10[/TD]
[TD]12[/TD]
[/TR]
[/TABLE]
شکل های قبلی با توضیح

[TR]
[TH]شکل های مارپیچ DNA[/TH]
[TH]حالت A[/TH]
[TH]حالتB[/TH]
[TH]حالت Z[/TH]
[/TR]
[TR]
[TD]نسبت های کلی[/TD]
[TD]کوتاه و پهن[/TD]
[TD]بزرگتر و باریکتر[/TD]
[TD]طویل و باریک[/TD]
[/TR]
[TR]
[TD]ارتفاع به ازای هرجفت باز[/TD]
[TD]3/2آنگستروم[/TD]
[TD]23/3آنگستروم[/TD]
[TD]8/3آنگستروم[/TD]
[/TR]
[TR]
[TD]قطر مارپیچ[/TD]
[TD]5/25آنگستروم[/TD]
[TD]7/23آنگستروم[/TD]
[TD]4/18آنگستروم[/TD]
[/TR]
[TR]
[TD]جهت چرخش مارپیچ[/TD]
[TD]راست گرد[/TD]
[TD]راست گرد[/TD]
[TD]چپ گرد[/TD]
[/TR]
[TR]
[TD]خمیدگی باز نسبت به محور مارپیچ[/TD]
[TD]19+[/TD]
[TD]2/1-[/TD]
[TD]9-[/TD]
[/TR]
[TR]
[TD]متوسط چرخش پروانه ای جفت باز[/TD]
[TD]18+[/TD]
[TD]16+[/TD]
[TD]حدود 0[/TD]
[/TR]
[TR]
[TD]موقعیت محور مارپیچ[/TD]
[TD]شیار بزرگ[/TD]
[TD]از میان جفت باز ها[/TD]
[TD]شیار کوچک[/TD]
[/TR]
[TR]
[TD]اندازه ی شیار بزرگ[/TD]
[TD]بسیار باریک با عمق زیاد[/TD]
[TD]پهن و عمق متوسط[/TD]
[TD]پهن شده به روی سطح مارپیچ[/TD]
[/TR]
[TR]
[TD]اندازه ی شیار کوچک[/TD]
[TD]بسیار پهن ولی کم عمق[/TD]
[TD]باریک و عمق متوسط[/TD]
[TD]بسیار باریک ولی خیلی عمیق[/TD]
[/TR]
[TR]
[TD]صورت بندی پیوند گلیکوزیدی[/TD]
[TD]آنتی[/TD]
[TD]آنتی[/TD]
[TD]آنتی در Cو سین در G[/TD]
[/TR]
[TR]
[TD]جفت باز در هر دور مارپیچ[/TD]
[TD]11[/TD]
[TD]10[/TD]
[TD]12[/TD]
[/TR]
[/TABLE]
پاسخ : ساختارهای dna
فکر نکنم استاد.
برای این نوع dna باید ترادف یک در میان پورین - پیریمیدین داشته باشیم، در ضمن z-dna در غلظت بالای نمک تشکیل میشه.
اگر هم جایی داشته باشیم-- مثلا در یک وزیکول-- شاید برای این باشه که سلول از روی آن dna به هیچ وجه همانند سازی نکنه.
آیا z-dna در حالت طبیعی سلول وجود دارد؟
برای این نوع dna باید ترادف یک در میان پورین - پیریمیدین داشته باشیم، در ضمن z-dna در غلظت بالای نمک تشکیل میشه.
اگر هم جایی داشته باشیم-- مثلا در یک وزیکول-- شاید برای این باشه که سلول از روی آن dna به هیچ وجه همانند سازی نکنه.
پاسخ : ساختارهای dna
چیزی که پیدا کردم و جالب بود :
ScienceDaily (Jan. 22, 2012) — New research published in BioMed Central's open access journal Cell & Bioscience is the first to show that left-handed Z-DNA, normally only found at sites where DNA is being copied, can also form on nucleosomes.
The structure of DNA which provides the blueprint for life has famously been described as a double helix. To save space inside the nucleus, DNA is tightly wound around proteins to form nucleosomes which are then further wound and compacted into chromatin, which is further compacted into chromosomes.
But this familiar image of a right handed coil (also called B-DNA) is not the only form of DNA. At sites where DNA is being copied into RNA (the messenger which is used as the instruction to make proteins) the DNA needs to unwind, and, in a process of negative supercoiling, can form a left-handed variety of the DNA double helix (Z-DNA).
It was originally thought that Z-DNA could only be formed in the presence of active RNA polymerase (the enzyme which assembles RNA). However more recently it has been discovered that SWI/SNF, a protein involved in remodeling nucleosomes and allowing RNA polymerase access to DNA, can convert certain sequences of B to Z-DNA.
The team of researchers led by Dr Keji Zhao discovered that they could convert B-DNA to Z-DNA on nucleosomes by the addition of SWI/SNF and ATP (the cell’s energy source) and that the Z-nucleosome formed was a novel structure.
Dr Zhao, from the NIH, explained, "The fact that we have found Z-DNA on nucleosomes is a new step in understanding the roles of chromosome remodeling and Z-DNA in regulating gene expression. While the Z-nucleosome is likely to be a transient structure it nevertheless provides a window of opportunity for the placement of DNA binding proteins which may recruit, regulate, or block the transcription machinery and hence protein expression."
اینم لینک:Elusive Z-DNA found on nucleosomes
چیزی که پیدا کردم و جالب بود :
ScienceDaily (Jan. 22, 2012) — New research published in BioMed Central's open access journal Cell & Bioscience is the first to show that left-handed Z-DNA, normally only found at sites where DNA is being copied, can also form on nucleosomes.
The structure of DNA which provides the blueprint for life has famously been described as a double helix. To save space inside the nucleus, DNA is tightly wound around proteins to form nucleosomes which are then further wound and compacted into chromatin, which is further compacted into chromosomes.
But this familiar image of a right handed coil (also called B-DNA) is not the only form of DNA. At sites where DNA is being copied into RNA (the messenger which is used as the instruction to make proteins) the DNA needs to unwind, and, in a process of negative supercoiling, can form a left-handed variety of the DNA double helix (Z-DNA).
It was originally thought that Z-DNA could only be formed in the presence of active RNA polymerase (the enzyme which assembles RNA). However more recently it has been discovered that SWI/SNF, a protein involved in remodeling nucleosomes and allowing RNA polymerase access to DNA, can convert certain sequences of B to Z-DNA.
The team of researchers led by Dr Keji Zhao discovered that they could convert B-DNA to Z-DNA on nucleosomes by the addition of SWI/SNF and ATP (the cell’s energy source) and that the Z-nucleosome formed was a novel structure.
Dr Zhao, from the NIH, explained, "The fact that we have found Z-DNA on nucleosomes is a new step in understanding the roles of chromosome remodeling and Z-DNA in regulating gene expression. While the Z-nucleosome is likely to be a transient structure it nevertheless provides a window of opportunity for the placement of DNA binding proteins which may recruit, regulate, or block the transcription machinery and hence protein expression."
اینم لینک:Elusive Z-DNA found on nucleosomes
پاسخ : ساختارهای dna
ترجمه ی متن بالا:
تحقیقات جدید علم (22، ژانویه 2012) - منتشر شده در ژورنال Cell BioMed مرکزی با دسترسی آزاد و افزار برای اولین بار نشان می دهد که دست چپ Z-DNA، به طور معمول تنها در سایت های که در آن DNA است که در حال کپی یافت می شود، همچنین می توانید در nucleosomes را تشکیل می دهنداست.
ساختار دی ان ای فراهم می کند که نوعی چاپ عکاسی که زمینه ان ابی ونقش ان سفید است برای زندگی مشهور شده است به عنوان یک مارپیچ دوگانه است. برای صرفه جویی در فضا در داخل هسته، DNA به صورت فشرده به دور پروتئین زخم به شکل nucleosomes که بیشتر زخم و فشرده را به کروماتین است که بیشتر به کروموزوم متراکم است.
اما این تصویر آشنا از یک سیم پیچ راست دست (همچنین به نام B-DNA) تنها فرم از DNA است. در سایت های که در آن DNA به RNA (پیامبر است که به عنوان دستور العمل را به پروتئین ها استفاده می شود) آن برداشتهاید. کارهای دارای حق انحصاری تکثیر DNA نیاز به باز کردن، و در روند از supercoiling منفی، می تواند انواع چپ دست مارپیچ دوگانه DNA (تشکیل می دهد Z-DNA).
در ابتدا تصور می شد که Z-DNA تنها می تواند در حضور RNA پلی مراز فعال (آنزیم که مونتاژ RNA) تشکیل شده است. با این حال اخیرا کشف شده است که SWI / SNF، پروتئین های درگیر در به بازسازی nucleosomes و اجازه دسترسی به RNA پلی مراز به DNA، توالی های خاصی از B به Z-DNA تبدیل.
تیم از محققان به رهبری دکتر Keji ژائو کشف کردند که آنها می توانند B-DNA Z-DNA در nucleosomes تبدیل توسط علاوه بر این SWI / SNF و ATP (منبع انرژی سلول) و تشکیل Z-نوکلئوزوم ساختار رمان بود.
دکتر Zhao، از NIH، توضیح داد: "این واقعیت که ما را به Z-DNA در nucleosomes یافت گام های جدید در فهم نقش بازسازی کروموزوم و Z-DNA در تنظیم بیان ژن است. در حالی که Z-نوکلئوزوم به احتمال زیاد ساختار گذرا اما با این وجود فراهم می کند یک پنجره فرصت برای قرار دادن پروتئین های اتصال DNA که ممکن است جذب، تنظیم، و یا بلوک ماشین آلات رونویسی و از این رو بیان پروتئین است. "
چیزی که پیدا کردم و جالب بود :
ScienceDaily (Jan. 22, 2012) — New research published in BioMed Central's open access journal Cell & Bioscience is the first to show that left-handed Z-DNA, normally only found at sites where DNA is being copied, can also form on nucleosomes.
The structure of DNA which provides the blueprint for life has famously been described as a double helix. To save space inside the nucleus, DNA is tightly wound around proteins to form nucleosomes which are then further wound and compacted into chromatin, which is further compacted into chromosomes.
But this familiar image of a right handed coil (also called B-DNA) is not the only form of DNA. At sites where DNA is being copied into RNA (the messenger which is used as the instruction to make proteins) the DNA needs to unwind, and, in a process of negative supercoiling, can form a left-handed variety of the DNA double helix (Z-DNA).
It was originally thought that Z-DNA could only be formed in the presence of active RNA polymerase (the enzyme which assembles RNA). However more recently it has been discovered that SWI/SNF, a protein involved in remodeling nucleosomes and allowing RNA polymerase access to DNA, can convert certain sequences of B to Z-DNA.
The team of researchers led by Dr Keji Zhao discovered that they could convert B-DNA to Z-DNA on nucleosomes by the addition of SWI/SNF and ATP (the cell’s energy source) and that the Z-nucleosome formed was a novel structure.
Dr Zhao, from the NIH, explained, "The fact that we have found Z-DNA on nucleosomes is a new step in understanding the roles of chromosome remodeling and Z-DNA in regulating gene expression. While the Z-nucleosome is likely to be a transient structure it nevertheless provides a window of opportunity for the placement of DNA binding proteins which may recruit, regulate, or block the transcription machinery and hence protein expression."
اینم لینک:Elusive Z-DNA found on nucleosomes
ScienceDaily (Jan. 22, 2012) — New research published in BioMed Central's open access journal Cell & Bioscience is the first to show that left-handed Z-DNA, normally only found at sites where DNA is being copied, can also form on nucleosomes.
The structure of DNA which provides the blueprint for life has famously been described as a double helix. To save space inside the nucleus, DNA is tightly wound around proteins to form nucleosomes which are then further wound and compacted into chromatin, which is further compacted into chromosomes.
But this familiar image of a right handed coil (also called B-DNA) is not the only form of DNA. At sites where DNA is being copied into RNA (the messenger which is used as the instruction to make proteins) the DNA needs to unwind, and, in a process of negative supercoiling, can form a left-handed variety of the DNA double helix (Z-DNA).
It was originally thought that Z-DNA could only be formed in the presence of active RNA polymerase (the enzyme which assembles RNA). However more recently it has been discovered that SWI/SNF, a protein involved in remodeling nucleosomes and allowing RNA polymerase access to DNA, can convert certain sequences of B to Z-DNA.
The team of researchers led by Dr Keji Zhao discovered that they could convert B-DNA to Z-DNA on nucleosomes by the addition of SWI/SNF and ATP (the cell’s energy source) and that the Z-nucleosome formed was a novel structure.
Dr Zhao, from the NIH, explained, "The fact that we have found Z-DNA on nucleosomes is a new step in understanding the roles of chromosome remodeling and Z-DNA in regulating gene expression. While the Z-nucleosome is likely to be a transient structure it nevertheless provides a window of opportunity for the placement of DNA binding proteins which may recruit, regulate, or block the transcription machinery and hence protein expression."
اینم لینک:Elusive Z-DNA found on nucleosomes
تحقیقات جدید علم (22، ژانویه 2012) - منتشر شده در ژورنال Cell BioMed مرکزی با دسترسی آزاد و افزار برای اولین بار نشان می دهد که دست چپ Z-DNA، به طور معمول تنها در سایت های که در آن DNA است که در حال کپی یافت می شود، همچنین می توانید در nucleosomes را تشکیل می دهنداست.
ساختار دی ان ای فراهم می کند که نوعی چاپ عکاسی که زمینه ان ابی ونقش ان سفید است برای زندگی مشهور شده است به عنوان یک مارپیچ دوگانه است. برای صرفه جویی در فضا در داخل هسته، DNA به صورت فشرده به دور پروتئین زخم به شکل nucleosomes که بیشتر زخم و فشرده را به کروماتین است که بیشتر به کروموزوم متراکم است.
اما این تصویر آشنا از یک سیم پیچ راست دست (همچنین به نام B-DNA) تنها فرم از DNA است. در سایت های که در آن DNA به RNA (پیامبر است که به عنوان دستور العمل را به پروتئین ها استفاده می شود) آن برداشتهاید. کارهای دارای حق انحصاری تکثیر DNA نیاز به باز کردن، و در روند از supercoiling منفی، می تواند انواع چپ دست مارپیچ دوگانه DNA (تشکیل می دهد Z-DNA).
در ابتدا تصور می شد که Z-DNA تنها می تواند در حضور RNA پلی مراز فعال (آنزیم که مونتاژ RNA) تشکیل شده است. با این حال اخیرا کشف شده است که SWI / SNF، پروتئین های درگیر در به بازسازی nucleosomes و اجازه دسترسی به RNA پلی مراز به DNA، توالی های خاصی از B به Z-DNA تبدیل.
تیم از محققان به رهبری دکتر Keji ژائو کشف کردند که آنها می توانند B-DNA Z-DNA در nucleosomes تبدیل توسط علاوه بر این SWI / SNF و ATP (منبع انرژی سلول) و تشکیل Z-نوکلئوزوم ساختار رمان بود.
دکتر Zhao، از NIH، توضیح داد: "این واقعیت که ما را به Z-DNA در nucleosomes یافت گام های جدید در فهم نقش بازسازی کروموزوم و Z-DNA در تنظیم بیان ژن است. در حالی که Z-نوکلئوزوم به احتمال زیاد ساختار گذرا اما با این وجود فراهم می کند یک پنجره فرصت برای قرار دادن پروتئین های اتصال DNA که ممکن است جذب، تنظیم، و یا بلوک ماشین آلات رونویسی و از این رو بیان پروتئین است. "
پاسخ : ساختارهای dna
اینم مقاله ی دوم:
البته این ابسترکتشه:
اینم مقاله ی دوم:
البته این ابسترکتشه:
Z-DNA forms transiently behind the active RNA polymerases, because of the mechanical torsional stress produced during transcription. In this paper, we explore the possibility that the distribution of Z-DNA stretches signals the sites related to nuclear transcription. To localize transcription, the in situ assay for active RNA polymerases, that allows the elongation of the already initiated transcripts but no initiation of new ones (run-on experiments), was carried out in isolated nuclei of Allium cepa L. root meristems. Both nucleolar and non-nucleolar sites appeared labelled. Nucleoli were most active in transcription than the multiple non-nucleolar foci altogether. In situ immunodetection of Z-DNA provided images that were comparable to those obtained after the run-on assay, with one exception: while Z-DNA and transcription sites were scattered throughout the whole nucleus, Z-DNA also accumulated in the nuclear periphery, where no transcription foci were detected in run-on assays. The peripheral Z-conformation signals might correspond to dsRNA segments present in the pre-mRNA in the process of their export to cytoplasm. The Z-containing structures nearly disappeared when non-nucleolar RNA polymerase II-dependent transcription had been previously abolished by the adenosine analogue DRB (5,6-dichloro-1-beta-D-ribofuranosylbenzimidazole). This inhibition selectively decreased the amount of all nucleoplasmic Z structures. On the other hand, the inhibition of the nucleolar RNA polymerase I by cordycepin (3'-deoxyadenosine) prevented the presence of Z-DNA in nucleoli. We propose to use the in situ immunodetection of Z-DNA as a marker of the transcription level in both nucleolus and non-peripheral nucleoplasmic regions of nuclei. Co-detection of Z-DNA and of intermediate filament (IF) proteins, the major components of the nuclear matrix, was also carried out. The IFA antibody recognizes a conserved epitope essential for dimerization of the multiple IF proteins. They co-localized with most nucleolar Z-DNA, but not with the nucleoplasmic ones. In the nuclear periphery, the Z-positive signals were adjacent to the IF proteins constituting the lamina, though both signals did not often co-localize.
اینم لینک:http://www.ncbi.nlm.nih.gov/pubmed/15145775
آخرین ویرایش توسط مدیر
پاسخ : ساختارهای dna
تا انجايي كه يادم مي ايد اركي باكتر ها و ترموفيل ها كه در شرايط سخت از جمله غلظت بالاي نمك و دمت زندگي مي كنند اين نوع dna وجود دارد .
فکر نکنم استاد.
برای این نوع dna باید ترادف یک در میان پورین - پیریمیدین داشته باشیم، در ضمن z-dna در غلظت بالای نمک تشکیل میشه.
اگر هم جایی داشته باشیم-- مثلا در یک وزیکول-- شاید برای این باشه که سلول از روی آن dna به هیچ وجه همانند سازی نکنه.
برای این نوع dna باید ترادف یک در میان پورین - پیریمیدین داشته باشیم، در ضمن z-dna در غلظت بالای نمک تشکیل میشه.
اگر هم جایی داشته باشیم-- مثلا در یک وزیکول-- شاید برای این باشه که سلول از روی آن dna به هیچ وجه همانند سازی نکنه.
پاسخ : ساختارهای dna
ساختار و ويژگي هاي هر كدام از اين DNA ها را كه كاربران گفتند. خودتان ايده بزنيد در كجاها ودرچه محيط هايي اين DNA هايافت مي شوند و سپس دنبال همچين محيطي براي زندگي جاندار بگردين .
مثلا همين Z-DNA چون پيوند سه گانه ي G-C زيادي در Syne Form تشكيل مي دهند مقاومت بيشتري نسبت به دناتوره شدن دربرابر حرارت و غلظت هاي بالاي نمك و .. دارند بنابراين بايد محيط شان داراي درجه حرارتي بالايي باشد + .. كه مي توان اين را در اركي باكتر ها (به صوص ترموفيل ها) كه راعماق اقيانوس در دماي 105' درجه سلسيوس به راحتي زندگي مي كنند ، ديد.
بقيه را هم با همين شيوه استدلال ايده بزنيد.
یه جمع بندی از اینکه هر کدوم ازونا در کجاها و در چه موقعیت هایی وجود دارند میشه ارائه بدین؟
مثلا همين Z-DNA چون پيوند سه گانه ي G-C زيادي در Syne Form تشكيل مي دهند مقاومت بيشتري نسبت به دناتوره شدن دربرابر حرارت و غلظت هاي بالاي نمك و .. دارند بنابراين بايد محيط شان داراي درجه حرارتي بالايي باشد + .. كه مي توان اين را در اركي باكتر ها (به صوص ترموفيل ها) كه راعماق اقيانوس در دماي 105' درجه سلسيوس به راحتي زندگي مي كنند ، ديد.
بقيه را هم با همين شيوه استدلال ايده بزنيد.